-
Cours :
- Premiers pas en Java (pdf)
- Classes et objets (pdf)
- Penser objet et encapsulation (pdf)
- Tests et enum (pdf)
- Égalité, final, static, surcharge et documentation (pdf)
- Types paramétrés et interfaces (pdf)
- Agrégation, composition, délégation et extension (pdf)
- Paquetage, accessibilité et exceptions (pdf)
- Final et notions avancées (pdf)
- Documents :
- TP :
Simulation de réseaux de régulation
Projet : Simulation de réseaux de régulation
On va travailler sur ce TP sur des réseaux de régulation, c’est-à-dire la simulation de l’expressivité des gènes et la concentration des protéines dans une cellule. Le projet est la continuation du TP 3. Néanmoins, les interactions seront beaucoup plus complexes, car la concentration d’une protéine pourra dans ce nouveau modèle impacter la production.
Consignes à suivre
Projet noté
Ce projet est à faire entre le 25 octobre et le 6 décembre (date des soutenances). Vous devez faire le projet seul. Il faudra que votre dépôt git soit à jour pour le 6 décembre à 8h. Toute mise à jour après cette date sera impossible. Le projet sera évalué et comptera pour 50% de la note finale.
Récupérer le dépôt
Comme pour le TP 2, on va utiliser git pour la gestion de versions. Il vous faut donc vous reporter aux consignes du TP 2. Le lien vers le projet à forker est le suivant : https://etulab.univ-amu.fr/alaboure/regulatory-network.
Exécuter le projet du dépôt
Pour compiler et exécuter votre programme, il faut passer par l’onglet gradle à droite.
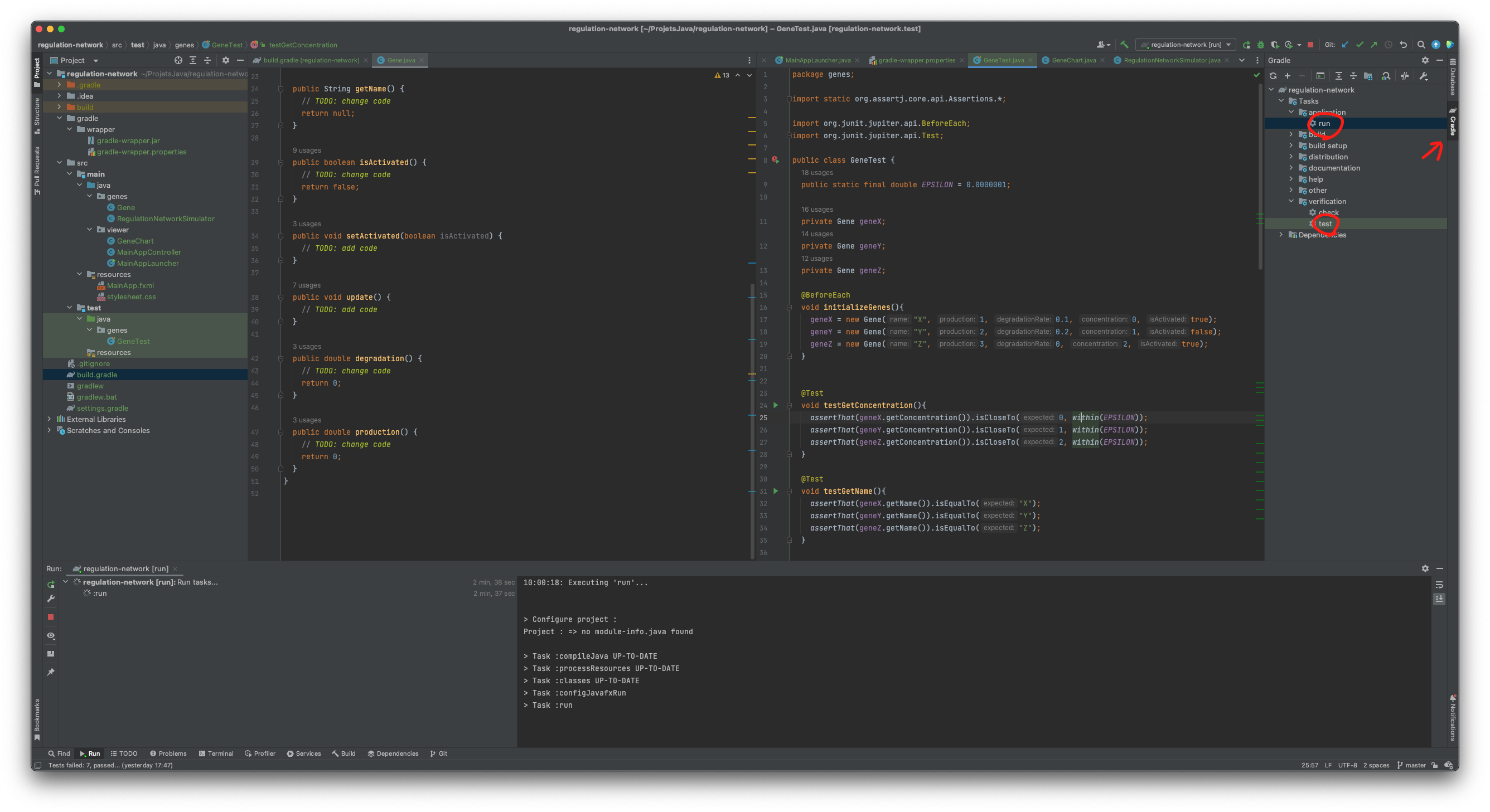
- Pour les tests, il faut cliquer deux fois sur
regulatory-network->Tasks->verification->test. Pour le moment, les tests ne passeront pas car certaines classes sont incomplètes. - Pour l’affichage, il faut cliquer deux fois sur
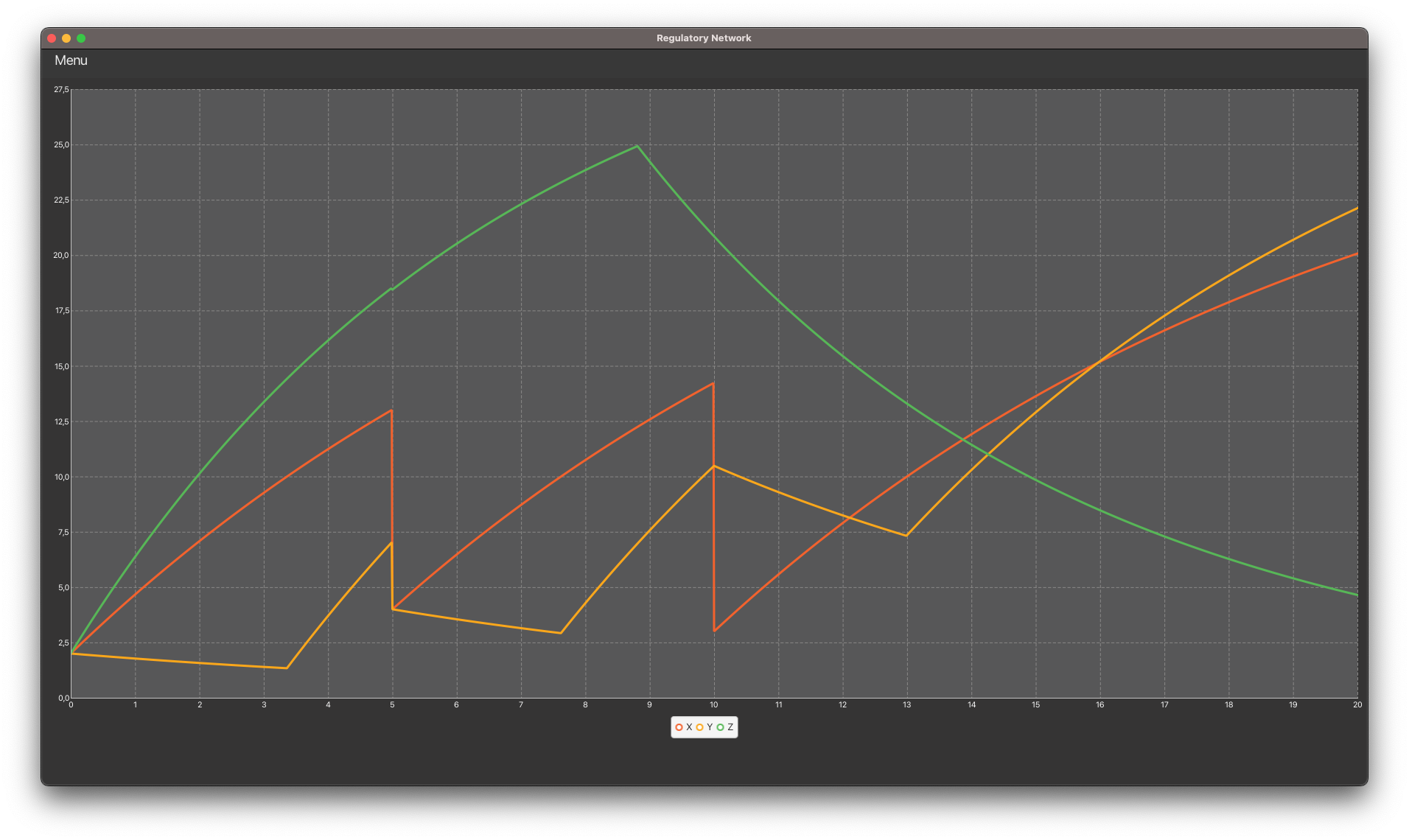
regulatory-network->Tasks->application->run. Vous devriez obtenir l’affichage suivant :
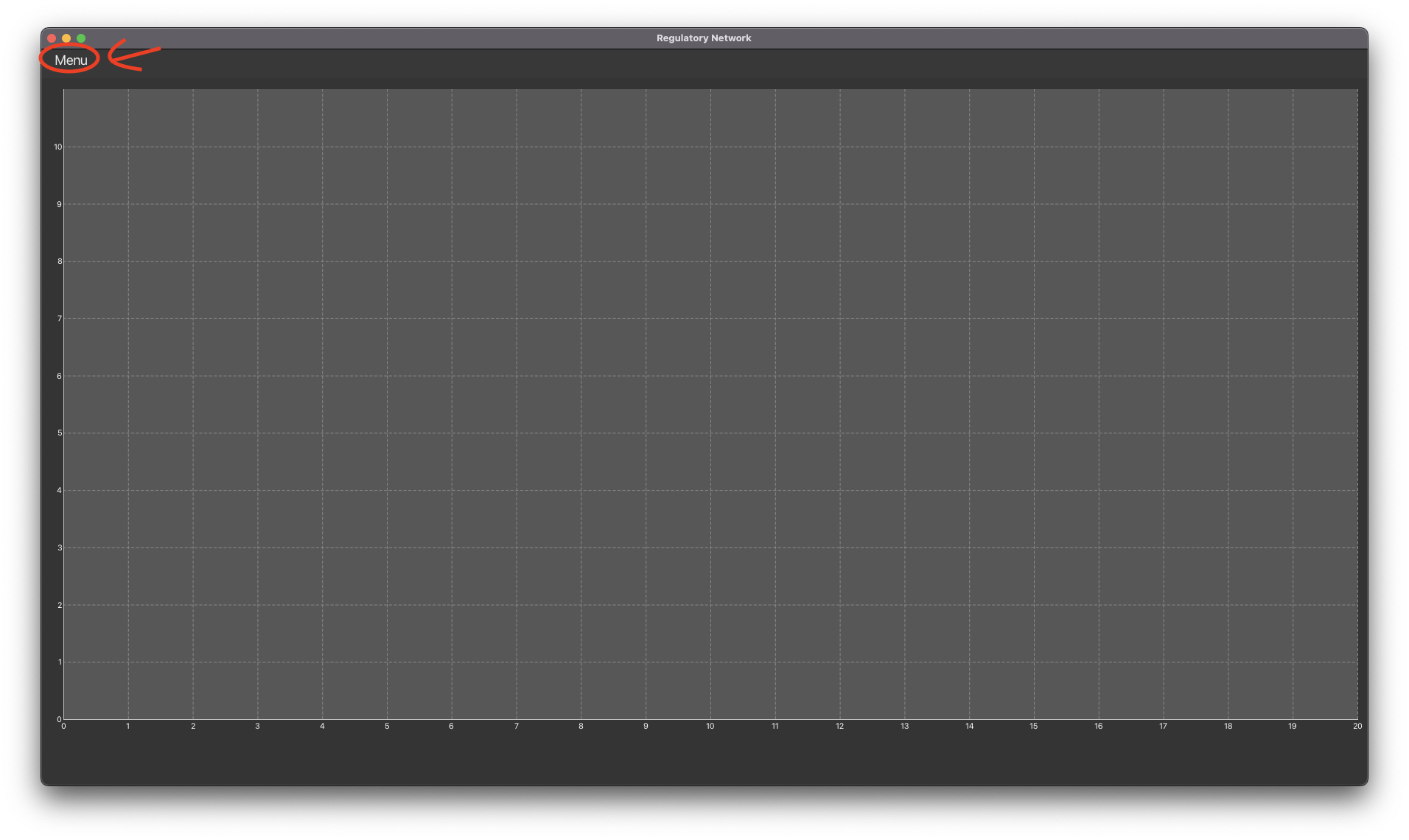
Le menu en à gauche vous permet d’accéder à trois fonctionnalités :
Generate data: génère des données à partir d’un réseau de régulation (RegulatoryNetwork) créé dans la méthodegeneratede la classemodel.network.RegulatoryNetworkDataManager;Open file: génère des données à partir d’un réseau de régulation (RegulatoryNetwork) défini dans un fichier d’extension.rgn(méthodereadde la classemodel.network.RegulatoryNetworkDataManager. Le projet contient des fichiers.rgnexemples dans le répertoiredatadu dépôt ;Write file: sauvegarde le réseau de régulation (RegulatoryNetwork) courant dans un fichier.rgn(méthodewritede la classemodel.network.RegulatoryNetworkDataManager).
Respect de la propriété intellectuelle
Nous vous demandons de ne pas partager votre programme, complet ou partiel, avec des membres d’autres équipes que la vôtre. Le non-respect de cette consigne vous expose à recevoir une note nulle. Tout emprunt que vous effectuez doit être proprement documenté en indiquant quelle partie de votre programme est concerné et de quelle source elle provient (nom d’un autre étudiant, site internet, etc.).
Critères d’évaluation
Vous serez évalué sur :
- La propreté du code : il est important de programmer proprement. Des répétitions de code trop visibles, des noms mal choisis ou des fonctions ayant beaucoup de lignes de code (plus de dix) vous pénaliseront. Le sujet vous donne les méthodes que vous devez absolument écrire mais il est tout à fait autoriser d’écrire des méthodes supplémentaires, de créer des constantes, … pour augmenter la lisibilité du code. On rappelle que vous devez écrire le code en anglais.
- La correction du code : on s’attend à ce que votre code soit correct, c’est-à-dire respecte les spécifications décrites dans le sujet. Vous avez tout intérêt à tester votre code pour vérifier son comportement.
- Modificateurs d’accès et attributs
final: dans ce sujet, on ne vous donnera pas toujours les modificateurs d’accès pour les différents éléments du code. Ce sera donc à vous de choisir l’accessibilité de certains éléments entre :private,public,protectedet default (pas de mot-clé). Vous aurez aussi à choisir si vous souhaitez utiliser le mot-cléfinalsur les attributs des classes. Vous serez évalué sur ces choix. - Les commit/push effectués : il vous faudra travailler en continu avec
gitet faire des push/commit le plus régulièrement possible. Un projet ayant très peu de push/commit effectués juste avant la date limite sera considéré comme suspicieux et noté en conséquence. Un minimum accepté pour ce projet sera d’au moins 2 pushs sur deux jours différents et d’au moins 10 commits au total. Si ces minimums ne sont pas respectés, le projet recevra une note de zéro. Vous devez faire un commit par méthode que vous codez et un push après chaque tâche.
Première modification de votre dépôt
Modifier le fichier README.md à la racine du projet. Ce fichier devra contenir votre nom de famille et votre prénom.
Ajout chargé de TP en tant que membre
Afin que je puisse évaluer votre code, il vous faudra m’en donner l’accès ne me rajoutant en tant que membre de votre dépôt.
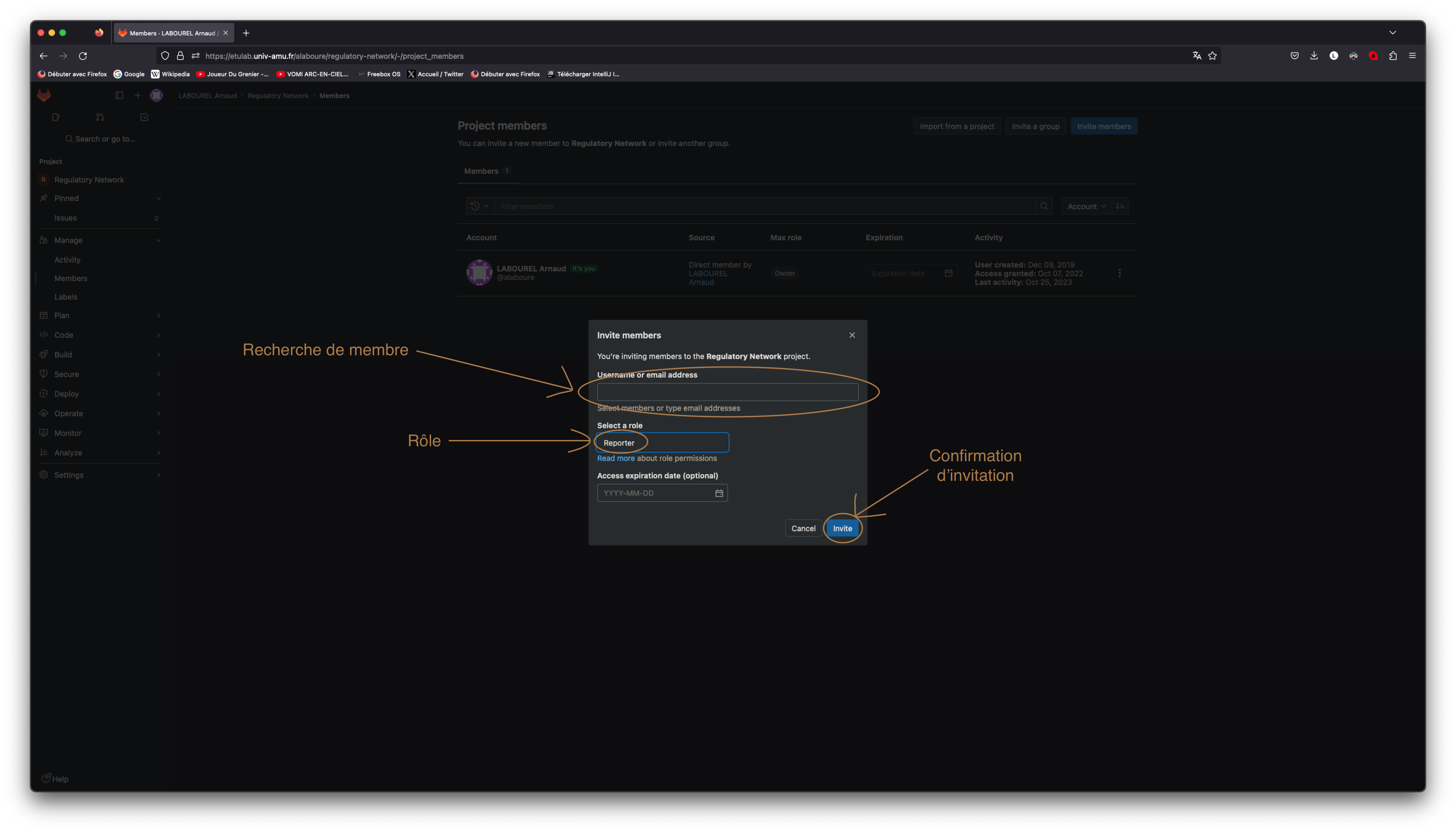
Une fois le projet créé (après le fork), vous devez me donner l’accès à votre code en cliquant sur Manage dans le menu de gauche puis members.
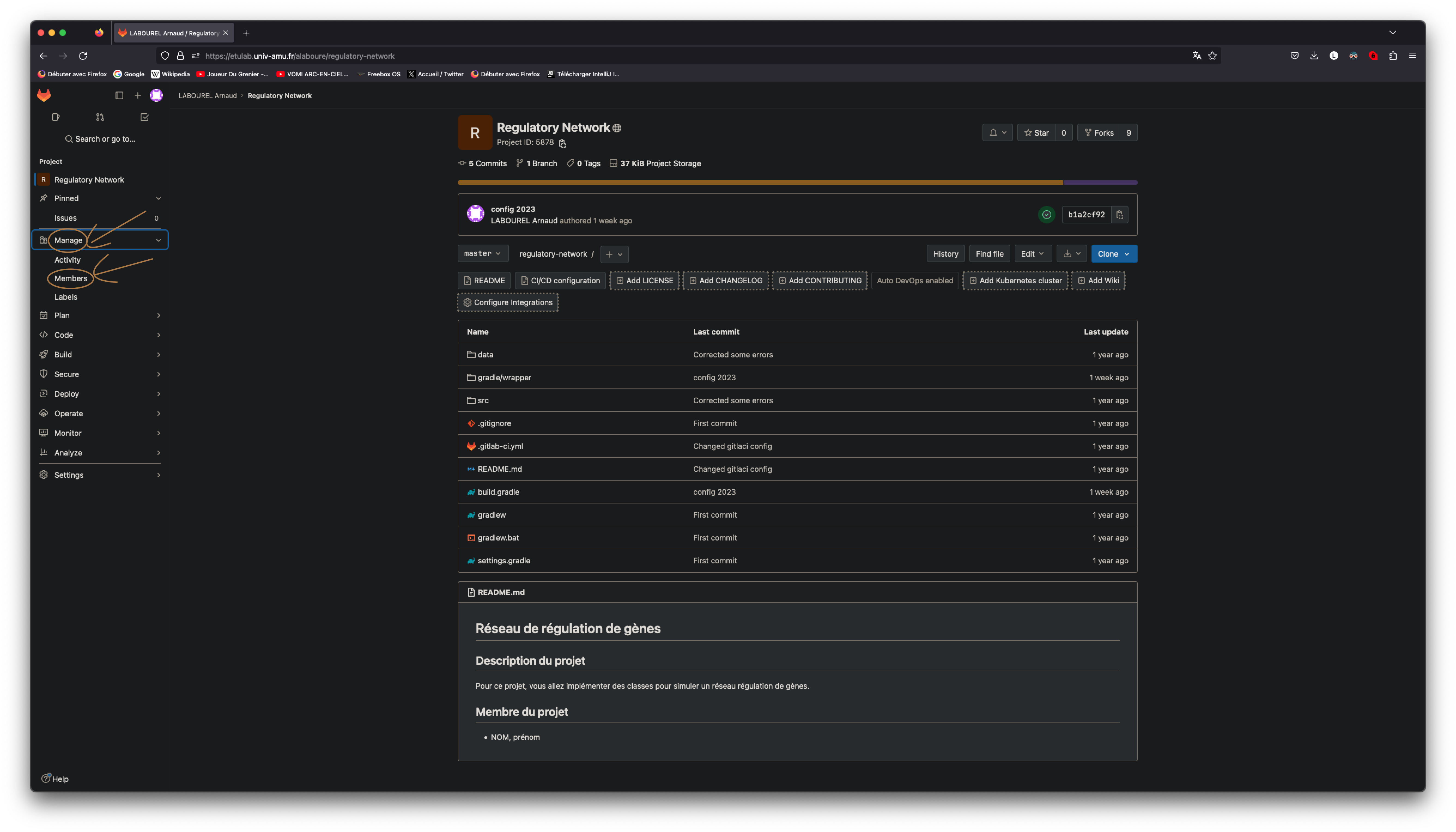
Une fois dans le menu de gestions des membres du projet, vous devez cliquer sur le bouton d’invitation en haut à gauche.
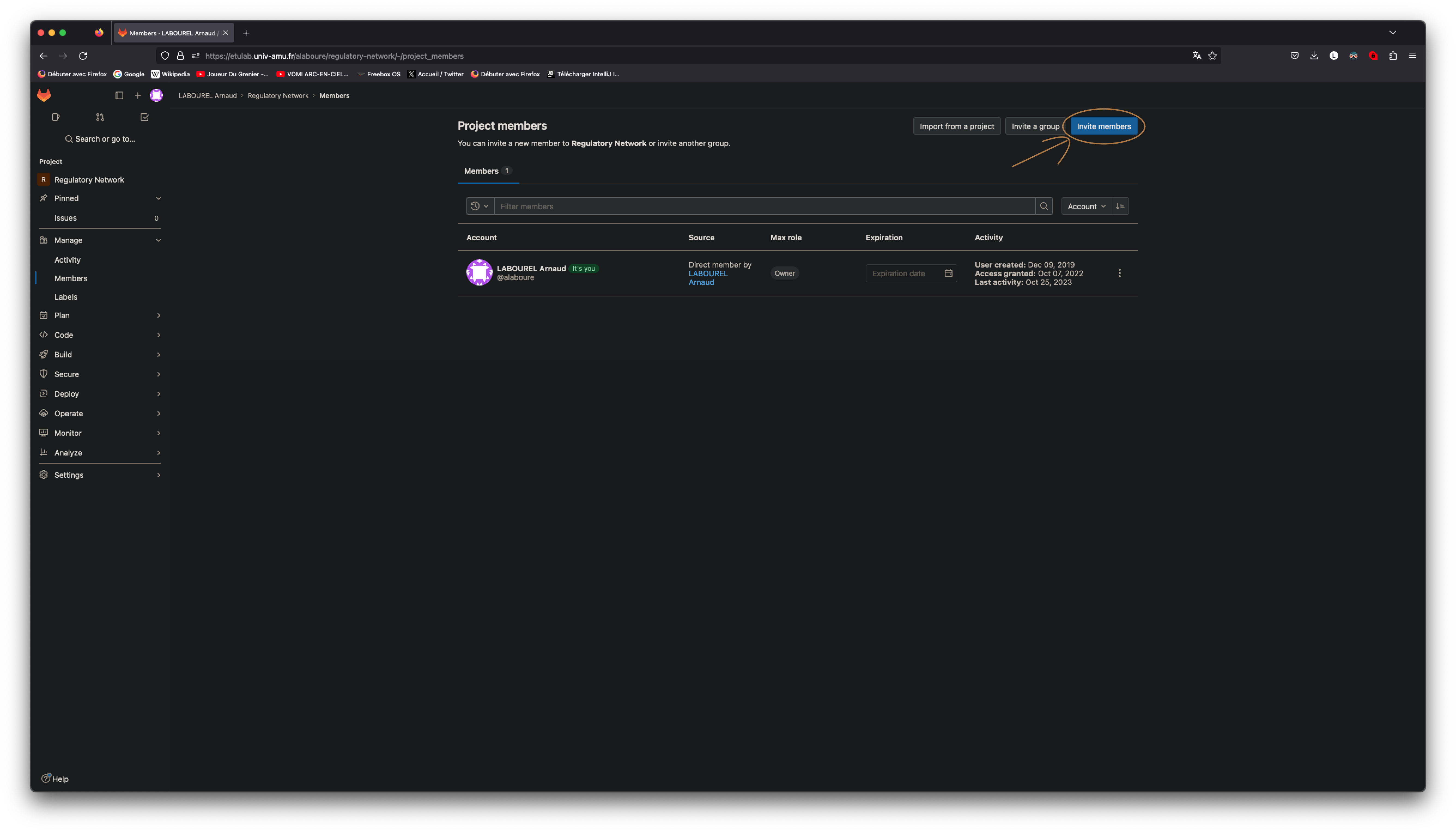
Ensuite vous pouvez me rechercher (login alaboure) dans la barre dédiée. Une fois que vous m’avez trouvé, vous pouvez me donner le rôle de reporter puis confirmer mon invitation en cliquant sur le bouton invite.
Simulation d’un gène
Explication de la simulation
La simulation est l’une des techniques fondamentales du calcul scientifique. Le programme contient un modèle de l’état du système et la manière de le mettre à jour après une étape de temps.
Pour ce projet, on va considérer la simulation de gènes dont chacun encode une protéine. Notre programme va simuler la concentration des protéines liées aux gènes considérées au sein d’une cellule au fil du temps.
Interface BasicGene
L’interface BasicGene correspond aux fonctionnalités basiques d’un gène. Elle contient les méthodes suivantes :
- une méthode
double getProteinConcentration()renvoie la concentration actuelle de la protéine ; - une méthode
double getInitialProteinConcentration()renvoie la concentration initiale (au moment de la création) de la protéine ; - une méthode
void setProteinConcentration(double proteinConcentration)fixe la concentration actuelle de la protéine ; - une méthode
String getName()qui renvoie le nom du gène (on supposera que chaque gène a un nom différent) ; - une méthode
void update(double duration)qui met à jour la concentration de la protéine en fonction de la production et de la dégradation de celle-ci, l’argumentdurationreprésente la durée de l’étape de temps et devra être utilisée pour calculer la nouvelle valeur de la concentration (par exemple sidurationvaut0.1alors on appliquera que 10% de la production et de la dégradation qui sont exprimées pour une unité de temps) ; - une méthode
double getMaximalProduction()qui renvoie la capacité de production maximale de la protéine pour une unité de temps ; - une méthode
double getDegradationRate()qui renvoie la proportion de la concentration qui disparaît à chaque unité de temps.
Interface RegulatedGene
L’interface RegulatedGene correspond à un gène régulé. Elle ajoute une propriété au gène : regulator.
L’attribut regulator contiendra l’objet modélisant le processus biologique régulant la production de la protéine du gène. Il permettra de calculer la production de la protéine en donnant la proportion (double compris entre 0 et 1) de la production maximale qui doit être produite. Cette proportion de production sera calculer par une méthode inputFunction du regulator.
L’interface RegulatedGene contient les méthodes suivantes (en plus de celles déjà présentes dans BasicGene) :
- Une méthode
Regulator getRegulator()qui renvoie le régulateur du gène ; - une méthode
void setRegulator(Regulator regulator)qui fixe le régulateur du gène ;
Interface RegulatoryGene
L’interface RegulatoryGene correspond à un gène pouvant réguler la production de protéine d’un autre gène. Elle ajoute une propriété au gène : isSignaled.
Le booléen isSignaled indique si la protéine produite par le gène peut influencer la production d’autres gènes. Le gène est dit signaled si certaines conditions sont remplies. Par exemple, certaines protéines ont un effet sur la production de protéines que si le niveau de sucre a atteint un certain seuil entraînant un changement de forme de la molécule qui permet l’interaction avec d’autres molécules. Dans notre cas, on changera depuis l’extérieur la propriété isSignaled à l’aide d’un setter.
L’interface RegulatedGene contient les méthodes suivantes (en plus de celles déjà présentes dans BasicGene) :
- une méthode
boolean isSignaled()qui renvoie la valeur deisSignaled; - une méthode
void setSignaled(boolean isSignaled)qui fixe la valeur deisSignaled.
Interface Gene
L’interface Gene correspond à un gène dans un réseau de régulation et donc pouvant à la fois être régulé, mais aussi participer à la régulation d’autres gènes.
Classe ConstantGene
Le code de cette classe vous est déjà donné. Elle permet de représenter un gène dont la concentration de la protéine ne change pas naturellement (pas de mise à jour de la concentration de la protéine avec la méthode update).
Interface Regulator
Le régulateur regulator est l’objet modélisant le processus biologique régulant la production de la protéine du gène. Il ne contient deux méthodes
double inputFunction()qui renvoie la proportion (doublecompris entre 0 et 1) de la production maximale qui doit être produite par le gène qu’il régule ;String description()qui affiche une description du régulateur avec ses informations à sauvegarder (utile pour l’écriture de fichier).
Travail à réaliser
Tâche 1 : classe AlwaysOnRegulator
Créer une classe
AlwaysOnRegulatordans le packagemodel.regulatorsqui implémente l’interfaceRegulatoret dont la méthodeinputFunctionrenvoie toujours1et dont la méthodedescriptionrenvoie une chaîne de caractères vide (ne contenant aucun caractère).
Tâche 2 : classe ConcreteGene
La classe ConcreteGene implémentera l’interface Gene et contiendra les attributs suivants :
- un régulateur (attribut
regulatorde typeRegulator) qui déterminera la proportion de la production de la protéine du gène, si celui-ci estnullon considérera que la protéine est produite avec la valeur maximale de production ; - une capacité de production maximale (attribut
maximalProductionde typedouble) qui sera la capacité maximale de production de la protéine pour une étape de temps ; - le taux de dégradation (attribut
degradationRatede typedouble) qui sera la proportion de la concentration qui disparaît à chaque étape de temps (appel àupdate) ; - la concentration initiale (à la construction du gène)
initialProteinConcentrationde la protéine dans la cellule ; - la concentration
proteinConcentrationde la protéine dans la cellule.
Créer la classe
ConcreteGenedans le packagemodel.genesqui implémente l’interfaceGene.
Les méthodes production() et degradation() calculent respectivement la production et la dégradation de la concentration de la protéine pour une unité de temps.
Pour tester la classe ConcreteGene, vous pouvez créer une classe de test dans le répertoire src/test/java. Vous pouvez aussi tester en modifiant le code generate de la classe model.network.RegulatoryNetworkDataManager afin d’ajouter des ConcreteGene dans le RegulatoryNetwork généré par la méthode. Vous pourrez ensuite afficher les données correspondantes via le menu avec Generate data.
Tâche 3 : classes régulateurs booléens
Une interaction classique entre gènes peut être représentée par des activateurs (activator) ou des répresseurs (repressor). L’idée est que la protéine d’un autre gène (ou bien du gène lui-même) peut avoir un effet bénéfique (activateur augmentant la production de la protéine) ou répressif (répresseur diminuant la production d’un gène) sur la production de la protéine. Une manière simple de modéliser cela est de considérer ce que l’on appellera des régulateurs booléens. Le régulateur s’activera qui si les deux conditions suivantes sont remplies :
- Le signal est présent pour le gène associé au régulateur (
isSignaleddu gène régulateur est àtrue). Biologiquement, le signal représente une condition pour que la régulation se fasse. Cela peut représenter des stimuli extérieurs comme la présence de sucre qui va affecter le processus de transcription en modifiant la forme de la protéine produite via le gène régulateur. - Un seuil (threshold) de concentration de la protéine produite par le gène régulateur est atteint, c’est-à-dire que la concentration de la protéine associée au régulateur est supérieure ou égale à une valeur fixée à la construction du régulateur.
Pour cette tâche, vous devez implémenter deux classes :
BooleanActivatordont l’inputFunctionrenvoie1si le gène régulateur est signalé et la concentration de la protéine du gène est supérieure ou égale à un seuil défini à la construction et0sinon.BooleanRepressordont l’inputFunctionrenvoie0si le gène régulateur est signalé et la concentration de la protéine du gène est supérieure ou égale à un seuil défini à la construction et1sinon.
Ces deux classes ayant beaucoup de points communs, on créera une classe abstraite BooleanRegulator qui sera héritée par les deux classes afin d’éviter la duplication de code. Cela nous donne le diagramme suivant :
La méthode description renverra une chaîne de caractère composée de la valeur du seuil et du nom du gène séparés par un espace.
Créer les classes
BooleanRegulator,BooleanRepressoretBooleanActivatordans le packagemodel.regulatorsen respectant le diagramme ci-dessus.
Tâche 4 : classes régulateurs composites
Parfois la régulation de la production d’une protéine ne dépend pas seulement d’un gène, mais de plusieurs gènes. Afin de modéliser cela, vous aller définir de nouveaux types de régulateurs qui seront une composition des régulateurs vus à la tâche précédente, c’est-à-dire qu’ils contiendront une liste regulators contenant des objets de type Regulator.
MaxCompositeRegulatordont l’inputFunctionrenvoie le maximum desinputFunctionde ses régulateurs.MinCompositeRegulatordont l’inputFunctionrenvoie le minimum desinputFunctionde ses régulateurs.
Ces deux classes ayant beaucoup de points communs, on créera une classe abstraite CompositeRegulator qui sera étendue par les deux classes afin d’éviter la duplication de code. Cela nous donne le diagramme suivant :
La méthode initialValue donne la valeur de base de la sortie d’inputFunction (0 ou 1) alors que cumulativeValue calcule l’opération à effectuer entre une valeur déjà calculée et un nouvel élément (max ou min).
La méthode description() renverra une chaîne de caractères représentant la liste des régulateurs (avec le nom de leur classe obtenu en appelant getClass().getSimpleName() sur le régulateur suivi de leur information obtenu en appelant description() sur le régulateur).
Créer les classes
CompositeRegulator,MinCompositeRegulatoretMaxCompositeRegulatordans le packagemodel.regulatorsen respectant le diagramme ci-dessus.
Tâche 5 : événements
Afin de pouvoir faire une simulation plus complexe, vous allez ajouter des événements dans la simulation. Les événements implémenteront l’interface SimulationEvent qui contiendra les 4 méthodes suivantes :
updateGenes: qui met à jour les gènes de l’événement ;getTime: qui donne le temps d’activation de l’événement ;getGenes: qui donne la liste des gènes concernés par l’événement ;description: qui donne une chaîne de caractère codant la valeur spécifique à l’événement (par exemple une concentration ou une valeur booléenne).
On considérera deux types d’événement :
SetProteinConcentrationEventqui met à jour les gènes concernés en leur changeant la concentration de leur protéine à une valeur définie à la construction ;SetSignaledEventqui met à jour les gènes concernés en leur changeant leur signal (valeur deisSignaled) à une valeur définie à la construction.
Ces deux classes ayant beaucoup de points communs, on créera une classe abstraite AbstractSimulationEvent qui sera héritée par les deux classes afin d’éviter la duplication de code. Cela nous donne le diagramme suivant :
Créer les classes
AbstractSimulationEvent,SetSignaledEventetSetProteinConcentrationEventdans le packagemodel.eventsen respectant le diagramme ci-dessus.
Tâche 6 : lecture/écriture de fichiers
Comme vous l’avez déjà vu, un format de fichier a été défini pour pouvoir sauvegarder un réseau de régulation (un instance de la classe network.RegulatoryNetwork), c’est-à-dire les gènes avec leurs interactions. Pour l’instant, le format ne permet que de gérer que les gènes de type ConstantGene. Dans l’exemple ci-dessous, chaque ligne correspond à un élément du réseau de régulation :
- La ligne 1 définit la valeur de
timeStepqui correspond à la fréquence de mise à jour de la simulation. Par exemple, dans cet exemple les gènes seront mis à jour (appel àupdate) tous les0.01unité de temps afin de calculer l’évolution des valeurs de concentration ; - La ligne 2 définit la valeur de
timeUpperBoundqui correspond à la valeur maximale de temps (la simulation va de 0 àtimeUpperBound) ; - Les lignes 3 à 5 correspondent chacune à un gène, chaque ligne commence par le type de l’objet suivi des valeurs pour l’instancier (le nom, la concentration initiale et la valeur de
isSignaled).
TimeStep 0.01
TimeUpperBound 20.0
ConstantGene X 3.0 true
ConstantGene Y 2.0 true
ConstantGene Z 4.0 falseModifier les méthodes
writeetreaddenetwork.RegulatoryNetworkDataManagerafin de pouvoir lire et écrire le fichier ci-dessous :
TimeStep 0.01
TimeUpperBound 20.0
ConcreteGene X 3.0 0.1 2.0 true
AlwaysOnRegulator X
ConcreteGene Y 4.0 0.12 2.0 true
BooleanActivator Y 10.0 X
ConcreteGene Z 5.0 0.15 2.0 true
BooleanRepressor Z 7.0 Y
SetProteinConcentrationEvent 10.0 X 3.0
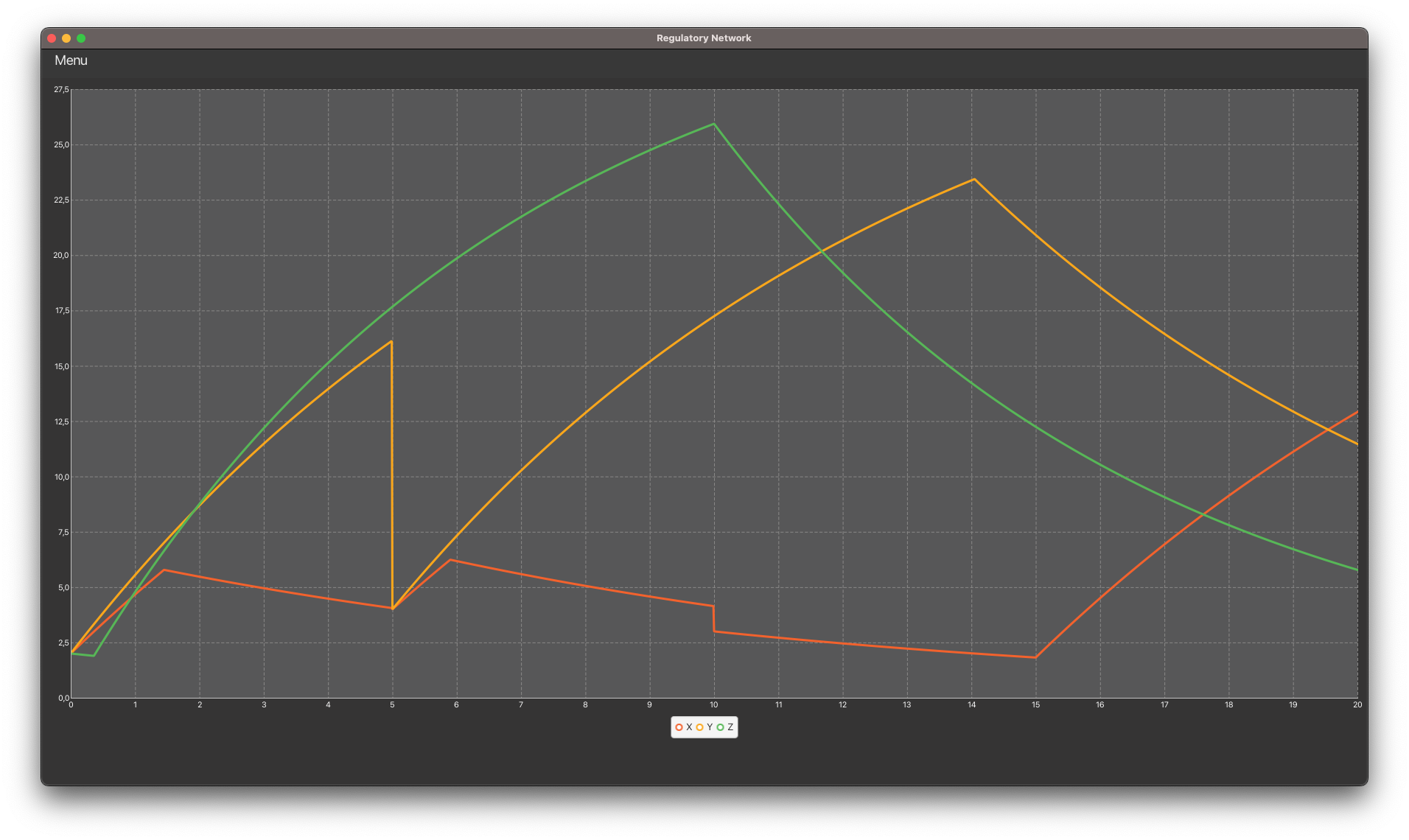
SetProteinConcentrationEvent 5.0 X,Y 4.0Ce fichier définit un RegulatoryNetwork équivalent à celui généré par le code ci-dessous :
public RegulatoryNetwork generate() {
List<Gene> genes = new ArrayList<>();
Gene x = new ConcreteGene("X", 3.0, 0.1, 2.0, true);
x.setRegulator(new AlwaysOnRegulator());
genes.add(x);
Gene y = new ConcreteGene("Y", 4.0, 0.12, 2.0, true);
genes.add(y);
y.setRegulator(new BooleanActivator(10, x));;
Gene z = new ConcreteGene("Z", 5.0, 0.15, 2.0, true);
genes.add(z);
z.setRegulator(new BooleanRepressor(7, y));
List<SimulationEvent> simulationEvents = new ArrayList<>();
simulationEvents.add(new SetProteinConcentrationEvent(List.of(x), 10.0, 3.0));
simulationEvents.add(new SetProteinConcentrationEvent(List.of(x, y), 5.0, 4.0));
return new RegulatoryNetwork(genes, simulationEvents, 0.01, 20.0);
}Vous devriez obtenir l’affichage suivant :
Tâche 7 : refactorisation du code pour l’écriture et la lecture de fichier (tâche difficile)
Pourquoi il faut changer le code
La manière dont on a programmé les fonctionnalités de lecture et d’écriture de fichier n’est pas totalement satisfaisante. En effet, la classe RegulatoryNetworkDataManager doit entièrement gérer ces deux fonctionnalités ce qui lui donne beaucoup de responsabilités. Cela contrevient au premier des principes SOLID connu sous le nom de principe de responsabilité unique (Single Responsibility Principle : SRP). Ce principe impose qu’une classe une classe, une fonction ou une méthode ne doit avoir qu’une et une seule responsabilité. Robert C. Martin qui a introduit ce principe l’exprime comme suivant : une classe ne doit changer que pour une seule raison (a class should have only one reason to change). Ici puisque la classe RegulatoryNetworkDataManager gère elle-même le format pour l’écriture et la lecture des différents types de gènes et de régulateurs ainsi que l’ordre des éléments dans le fichier, elle a plusieurs raisons de changer (pour l’ajout d’un nouveau type de gène, de régulateurs ou bien en changement de codage du fichier utilisant par exemple le XML). On va donc résoudre cette problématique en refactorant (réécrivant) le code pour les fonctionnalités de lecture et écriture de fichier afin que les responsabilités soient réparties entre plusieurs classes.
On va tout d’abord créer une interface EntitySerializer qui va nous permettre de regrouper, pour chaque type d’objet, son écriture (sérialisation) et sa lecture (désérialisation) dans une même classe.
L’interface EntitySerializer<E> est une interface paramétrée par un type E qui correspond au type de l’objet à sérialiser ou désérialiser. Cette interface contient les trois méthodes suivantes :
getCode: qui renvoie un identifiant de l’objet à sérialiser ou désérialiser, c’est-à-dire généralement le nom de sa classe ;serialize: retourne une chaîne qui décrit l’objet à sérialiser ;deserialize: reconstruit un objet à partir d’une chaîne de caractères qui le décrit (généralement une ligne du fichier qui a été produite par la méthodeserialize).
Plus précisément, l’interface est la suivante :
public interface EntitySerializer<E> {
String getCode();
String serialize(E entity, RegulatoryNetworkWriter writer);
E deserialize(String string, RegulatoryNetworkReader reader);
}La classe RegulatoryNetworkWriter correspond à un objet qui va nous permettre d’écrire dans un fichier et aura donc une méthode void write(BufferedWriter bufferedWriter, RegulatoryNetwork regulatoryNetwork) alors que la classe correspond à un objet qui va nous permettre de lire un fichier et aura donc une méthode RegulatoryNetwork read(BufferedReader bufferedReader). Avoir accès à ses objets dans les méthodes de sérialisation et désérialisation sera utile par la suite. Pour le moment, vous pouvez vous contenter de créer des classes quasi-vides pour ces deux types d’objets.
Lecture/écriture de gènes
Vous allez commencer par coder les classes permettant de créer un réseau de régulation ne contenant que des gènes (pas d’événements ni de régulateurs pour le moment). Pour cela, vous allez tout d’abord coder les classe de sérialisations pour les deux types de gènes. La classe ConcreteGeneSerializer devra implémenter l’interface EntitySerializer<ConcreteGene> et la classe ConstantGeneSerializer devra implémenter l’interface EntitySerializer<ConstantGene>.
Les deux classes devront aussi utiliser le patron de conception singleton. Ce patron garantit que l’instance d’une classe n’existe qu’en un seul exemplaire, tout en fournissant un point d’accès global à cette instance. Cela permet pour chaque classe implémentant EntitySerializer<E> de donner l’accès à une instance de leur classe grâce à leur méthode de classe getInstance.
Créer les classes
ConcreteGeneSerializeretConstantGeneSerializerdans le packagemodel.file.serializers.geneen respectant le diagramme ci-dessus.
Maintenant, il va falloir maintenant réécrire la partie lecture et la partie écriture du fichier dans votre code en repartant d’une version qui n’écrira que les gènes du réseau de régulation (pas les événements ni les régulateurs).
Vous allez commencer par partie écriture de fichier qui sera effectuer par la classe RegulatoryNetworkWriter. Pour cela, vous allez utiliser le patron de conception visitor qui permet de séparer une opération sur des objets et les objets sur lesquels on souhaite appliquer l’opération. Dans notre cas le diagramme de classe est le diagramme ci-dessous :
Les méthodes accept que vous devrez implémenter dans les deux types de gènes et rajouter dans l’interface Gene se contente d’appeler la méthode visit du visiteur sur l’objet courant (le this). Cela peut sembler étrange de créer une telle méthode, mais l’appel à accept permet d’appeler la bonne méthode visit. En effet, si on a une variable de type Gene, on ne sait pas si l’objet qu’elle contient est de type ConstantGene ou bien ConcreteGene. Appeler accept sur cet objet va donc appeler la bonne version de visit qui dépendra du type réel de l’objet.
C’est donc les méthodes visit qui devront retourner la chaîne de caractères à renvoyer. Pour cela, il suffira de récupérer (grâce à getInstance) le sérialiseur correspondant au type de l’objet en argument de visit et d’appeler la méthode serialize.
Créer l’interface
GeneVisitoret la classeConcreteGeneVisitordans le packagemodel.file.writeren respectant les consignes ci-dessus. Vous devrez aussi modifier ’interfaceGeneet les classesConcreteGeneetConstantGene.
Vous allez maintenant coder la classe RegulatoryNetworkWriter suivant le diagramme ci-dessous.
La méthode writeGenes devra écrire les gènes en utilisant le visiteur geneVisitor alors que writeConfiguration écrira les deux premières lignes du fichier codant les valeurs de TimeStep et TimeUpperBound.
Créer la classe
RegulatoryNetworkWriterdans le packagemodel.file.writeren respectant les consignes ci-dessus et testez là.
Il reste maintenant à coder la partie lecture de fichier. Vous allez donc coder la classe RegulatoryNetworkWriter suivant le diagramme ci-dessous.
La classe RegulatoryNetworkReader va permettre de lire un fichier et de produire un RegulatoryNetwork grâce à la méthode read.
Les Map sont des dictionnaires de java que vous pourrez initialiser avec des instances de HashMap. La classe RegulatoryNetworkReader contient les deux attributs de type Map suivants :
geneSerializerscontient les sérialiseurs des deux types de gènes qui sont stockés avec comme clé le code des objets qui sérialise (donné par la méthodegetCode). Cela permettra lorsqu’on lira le fichier de retrouver facilement le bon sérialiseur à partir du premier mot de la ligne. Les sérialiseurs de gènes sont de typeEntitySerializer<? extends Gene>qui correspond au typeEntitySerializeravec comme paramètre de type, un type inconnu?qui est une sous-classe deGene, c’est-à-dire qui étend ou implémenteGene).genescontient les gènes lus dans le fichier (ajouter après la lecture d’une ligne dans la méthoderead) qui sont stockés avec comme clé le nom du gène.
La classe RegulatoryNetworkReader contient les méthodes suivantes :
addGeneetgetGenequi permettent d’accéder aux gènes contenus dansgenes;addGeneSerializeretgetGeneSerializerqui permettent d’accéder aux sérialiseurs de gènes contenus dansgeneSerializers;- un constructeur qui va initialiser
geneSerializersen y ajoutant les deux types de sérialiseurs pour les gènes ; readqui va initialisergenes, lire les lignes du fichier correspondant aubufferedReaderen ajoutant les gènes décrits dans le fichier dansgeneset construire unRegulatoryNetworkpour le renvoyer.
Créer la classe
RegulatoryNetworkReaderdans le packagemodel.file.readeren respectant les consignes ci-dessus et testez là.
Lecture/écriture d’événements
Maintenant vous allez devoir vous attaquer à la lecture et écriture d’événement. Pour cela, il va vous falloir faire les modifications suivantes.
- Modifiez l’interface
RegulatoryGeneet les classes l’implémentant de sort à rajouter une méthodeboolean getInitialIsSignaled()qui renvoie la valeur donnée à la construction de gène pour l’attributisSignaled. Cette méthode sera à utiliser dans la sérialisation du gène afin d’écrire la valeur initiale deisSignaled(celle à la création de l’objet) et non la valeur courante qui a pu changer avec la simulation. - Créez une classe
ListGeneSerializerimplémentantEntitySerializer<List<Gene>>dans le packagemodel.file.serializers.list. Cette classe devra par exemple créer une liste de trois gènes dont les noms sontX,YetZà partir de la chaîne de caractères"[X,Y,Z]"et réaliser l’opération inverse, c’est-à-dire produire une chaîne de caractères"[X,Y,Z]"à partir de la liste des trois gènes. - Créez une classe
SetProteinConcentrationEventSerializerimplémentantEntitySerializer<SetProteinConcentrationEvent>et une classeSetSignaledEventSerializerimplémentantEntitySerializer<SetSignaledEvent>dans le packagemodel.file.serializers.event. - Créez une interface
EventVisitoret une classeConcreteEventVisitorl’implémentant dans le packagefile.writer. - Modifiez
SimulationEvent,SetSignaledEventetSetProteinConcentrationEventafin qu’elles acceptent (méthodeString accept(EventVisitor eventVisitor)) un visiteur. - Modifiez
RegulatoryNetworkWriteren ajoutant un attribut de typeEventVisitoret une méthodewriteEventsqui sera appelée dans la méthodewritepour écrire les événements. - Modifiez
RegulatoryNetworkReaderen ajoutant un attribut de typeMap<String, EntitySerializer<? extends SimulationEvent>>et un attribut de typeList<SimulationEvent>ainsi qu’en modifiant la méthodereadpour lire des événements pour prendre en compte les événements.
Lecture/écriture de régulateurs
- Créez une classe
ListRegulatorSerializerimplémentantEntitySerializer<List<Regulator>>dans le packagemodel.file.serializers.list. Cette classe devra par exemple créer une liste de deux régulateurs à partir de la chaîne de caractères"[BooleanRepressor 2.0 Z,BooleanActivator 2.0 X]"et réaliser l’opération inverse, c’est-à-dire produire une chaîne de caractères"[BooleanRepressor 2.0 Z,BooleanActivator 2.0 X]]"à partir de la liste des deux régulateurs. On supposera que les régulateurs de la liste ne sont pas des régulateurs composites. - Créez les classes suivantes dans le package
model.file.serializers.regulator:AlwaysOffRegulatorSerializerimplémentantEntitySerializer<AlwaysOffRegulator>AlwaysOnRegulatorSerializerimplémentantEntitySerializer<AlwaysOnRegulator>BooleanActivatorSerializerimplémentantEntitySerializer<BooleanActivator>BooleanRepressorSerializerimplémentantEntitySerializer<BooleanRepressor>MaxCompositeRegulatorSerializerimplémentantEntitySerializer<MaxCompositeRegulator>MinCompositeRegulatorSerializerimplémentantEntitySerializer<MinCompositeRegulator>
- Créez une interface
RegulatorVisitoret une classeConcreteRegulatorVisitorl’implémentant dans le packagefile.writer. - Modifiez
AlwaysOffRegulator,AlwaysOnRegulator,BooleanActivator,BooleanRepressor,BooleanRepressor,MaxCompositeRegulatoretMinCompositeRegulatorafin qu’elles acceptent (méthodeString accept(RegulatorVisitor regulatorVisitor)) un visiteur. - Modifiez
RegulatoryNetworkWriteren ajoutant un attribut de typeRegulatorVisitoret une méthodewriteRegulatorsqui sera appelée dans la méthodewritepour écrire les régulateurs. On va changer le format de fichier pour les régulateurs en écrivant d’abord le nom du gène sur lequel le régulateur s’applique puis le type de régulateur. - Modifiez
RegulatoryNetworkReaderen ajoutant un attribut de typeMap<String, EntitySerializer<? extends Regulator>>ainsi qu’en modifiant la méthodereadpour prendre en compte les régulateurs.
Faites les modifications demandées ci-dessus afin de pouvoir lire et écrire le fichier ci-dessous :
TimeStep 0.01
TimeUpperBound 20.0
ConcreteGene X 2.0 3.0 0.1 true
ConcreteGene Y 2.0 4.0 0.12 true
ConcreteGene Z 2.0 5.0 0.15 true
X MaxCompositeRegulator [BooleanRepressor 7.0 Y,BooleanRepressor 2.0 Y]
Y MaxCompositeRegulator [BooleanRepressor 2.0 Z,BooleanActivator 2.0 X]
Z MinCompositeRegulator [BooleanActivator 3.0 X,BooleanActivator 1.0 Z]
SetProteinConcentration 10.0 [X] 3.0
SetProteinConcentration 5.0 [X,Y] 4.0
SetSignaledEvent 15.0 [X,Y] falseCe fichier définit un RegulatoryNetwork équivalent à celui généré par le code ci-dessous :
public RegulatoryNetwork generate() {
List<Gene> genes = new ArrayList<>();
Gene x = new ConcreteGene("X", 3.0, 0.1, 2.0, true);
genes.add(x);
Gene y = new ConcreteGene("Y", 4.0, 0.12, 2.0, true);
genes.add(y);
Gene z = new ConcreteGene("Z", 5.0, 0.15, 2.0, true);
genes.add(z);
BooleanActivator booleanActivator1 = new BooleanActivator(3, x);
BooleanRepressor booleanRepressor1 = new BooleanRepressor(7, y);
BooleanActivator booleanActivator2 = new BooleanActivator(1, z);
BooleanRepressor booleanRepressor2 = new BooleanRepressor(2, y);
BooleanActivator booleanActivator3 = new BooleanActivator(2, x);
BooleanRepressor booleanRepressor3 = new BooleanRepressor(2, z);
MinCompositeRegulator minCompositeRegulator =
new MinCompositeRegulator(List.of(booleanActivator1,booleanActivator2));
MaxCompositeRegulator maxCompositeRegulator1 =
new MaxCompositeRegulator(List.of(booleanRepressor1,booleanRepressor2));
MaxCompositeRegulator maxCompositeRegulator2 =
new MaxCompositeRegulator(List.of(booleanRepressor3,booleanActivator3));
x.setRegulator(maxCompositeRegulator1);
y.setRegulator(maxCompositeRegulator2);
z.setRegulator(minCompositeRegulator);
List<SimulationEvent> simulationEvents = new ArrayList<>();
simulationEvents.add(new SetProteinConcentrationEvent(List.of(x),10.0,3.0));
simulationEvents.add(new SetProteinConcentrationEvent(List.of(x,y),5.0,4.0));
simulationEvents.add(new SetSignaledEvent(List.of(x, y),15.0,false));
return new RegulatoryNetwork(genes, simulationEvents, 0.01, 20.0);
}Vous devriez obtenir l’affichage suivant :
Tâche 8 : fonctionnalités additionnelles
Vous pouvez ajouter des fonctionnalités supplémentaires au simulateur, comme les suivantes :
Ajouter des classes pour définir des régulateurs de Hill qui sont plus fin que les régulateurs booléens :
- l’activateur de Hill utilise comme
inputFunctionla formule ci-dessous :
\[f(c) = \frac{c^n}{k^n+c^n}\]
avec \(c\) la concentration de la protéine du régulateur, \(k\) le coefficient d’activation et \(n\) le coefficient de Hill.
- le répresseur utilise comme
inputFunctionla formule ci-dessous :
\[f(c) = \frac{1}{1 +\left(\frac{c}{k}\right)^n}\]
avec \(c\) la concentration de la protéine du régulateur, \(k\) le coefficient d’activation et \(n\) le coefficient de Hill.
- l’activateur de Hill utilise comme
Ajouter des levées d’exception de type
IllegalArgumentExceptionlorsqu’une personne essaye d’appeler des constructeurs ou des méthodes avec des valeurs incorrectes (comme une valeur de concentration strictement négative ou une proportion strictement supérieure à 1).Ajouter les régulateurs composites dans la lecture/écriture de fichier.
Ajouter une fonctionnalité permettant de sauvegarder les données produites pour afficher les courbes (valeur de concentration des protéines) dans un fichier gnuplot.
Modifiez votre code pour gérer en lecture et écriture de fichier
Ajouter les fonctionnalités de votre choix pour rendre la simulation plus réaliste.